BCUT記述子
化学構造は、しばしば「グラフ」というデータ構造として取り扱われます。
このとき、各原子はノード(点)、原子間の結合はエッジ(線)と呼ばれ、
エッジの連結情報は、隣接行列として表現できます。
Burden*1は、この隣接行列の対角成分に各原子の属性を格納し、
化学構造情報をひとつの行列Bとして集約しようとしました。
- 構成原子に番号 1...n をふる。ただし、水素原子は省略する
- 対角成分には、対応する原子番号を入れる
- 一重、二重、三重、芳香性結合に対応する要素には、それぞれ0.1, 0.2, 0.3, 0.15を入れる
- 末端原子に関連する要素には、0.01を加える
- それ以外の要素は、0.001とする
こうして作られたBurden行列Bの固有値は、線形代数学の観点から
化学構造を数値(ベクトル)に畳み込んだものだと考えることができます。
CDKでは、原子番号の代わりに、原子量(B1)、部分電荷(B2)、極性(B3)を対角成分に代入して、
それぞれ固有値を計算しています。
たとえば、酢酸(CH3COOH)を原子量で計算した場合、下図のようになります。
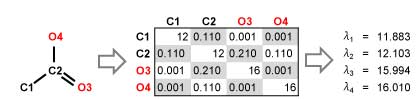
それでは、原著に倣い、固有値のうち、最小値(λ1)と最大値(λn)を出力してみましょう。
(化合物ID, B1最小固有値, B1最大固有値, B2最小固有値, B2最大固有値, B3最小固有値, B3最大固有値)
/* bcut.java */import java.io.*;
import org.openscience.cdk.interfaces.*;
import org.openscience.cdk.io.iterator.IteratingSMILESReader;
import org.openscience.cdk.interfaces.IMolecule;
import org.openscience.cdk.tools.manipulator.AtomContainerManipulator;import Jama.EigenvalueDecomposition;
import Jama.Matrix;
import org.openscience.cdk.CDKConstants;
import org.openscience.cdk.Molecule;
import org.openscience.cdk.annotations.TestClass;
import org.openscience.cdk.annotations.TestMethod;
import org.openscience.cdk.aromaticity.CDKHueckelAromaticityDetector;
import org.openscience.cdk.charges.GasteigerMarsiliPartialCharges;
import org.openscience.cdk.charges.GasteigerPEPEPartialCharges;
import org.openscience.cdk.charges.Polarizability;
import org.openscience.cdk.config.IsotopeFactory;
import org.openscience.cdk.exception.CDKException;
import org.openscience.cdk.graph.PathTools;
import org.openscience.cdk.graph.matrix.AdjacencyMatrix;
import org.openscience.cdk.interfaces.IAtomContainer;
import org.openscience.cdk.interfaces.IBond;
import org.openscience.cdk.qsar.DescriptorSpecification;
import org.openscience.cdk.qsar.DescriptorValue;
import org.openscience.cdk.qsar.IMolecularDescriptor;
import org.openscience.cdk.qsar.result.DoubleArrayResult;
import org.openscience.cdk.qsar.result.DoubleArrayResultType;
import org.openscience.cdk.qsar.result.IDescriptorResult;
import org.openscience.cdk.tools.CDKHydrogenAdder;
import org.openscience.cdk.tools.LoggingTool;
import org.openscience.cdk.tools.LonePairElectronChecker;
import org.openscience.cdk.tools.manipulator.AtomContainerManipulator;class bcut {
public static void main(String args[]){if(args.length!=1){
System.err.println("bcut <smiles-file>");
System.exit(1);
}
FileInputStream fis = null;
IteratingSMILESReader isr = null;
try{
fis = new FileInputStream(new File(args[0]));
isr = new IteratingSMILESReader(fis);
} catch (Exception e) {
e.printStackTrace();
}int nhigh = 1;
int nlow = 1;
boolean checkAromaticity = true;while( isr.hasNext() ){
Molecule mol = (Molecule)isr.next();
String id = (String)mol.getProperty("cdk:Title");int nheavy = 0;
int counter;
try{
// add H's in case they're not present
AtomContainerManipulator.percieveAtomTypesAndConfigureAtoms(mol);
CDKHydrogenAdder hAdder = CDKHydrogenAdder.getInstance(mol.getBuilder());
hAdder.addImplicitHydrogens(mol);
AtomContainerManipulator.convertImplicitToExplicitHydrogens(mol);
if ( checkAromaticity ) {
AtomContainerManipulator.percieveAtomTypesAndConfigureAtoms(mol);
}
CDKHueckelAromaticityDetector.detectAromaticity(mol);// find number of heavy atoms
for (int i = 0; i < mol.getAtomCount(); i++) {
if ( ! mol.getAtom(i).getSymbol().equals("H") ) nheavy++;
}
} catch (Exception e1) {
e1.printStackTrace();
}if (nheavy == 0) continue;
double[] diagvalue = new double[nheavy];
double[] eval1 = new double[nheavy];
double[] eval2 = new double[nheavy];
double[] eval3 = new double[nheavy];try {
// get atomic mass weighted BCUT
counter = 0;
for (int i = 0; i < mol.getAtomCount(); i++) {
if ( mol.getAtom(i).getSymbol().equals("H") ) continue;
diagvalue[counter] = IsotopeFactory.getInstance(mol.getBuilder()).
getMajorIsotope(mol.getAtom(i).getSymbol()).getExactMass();
counter++;
}
double[][] burdenMatrix = BurdenMatrix.evalMatrix(mol, diagvalue);
Matrix matrix = new Matrix(burdenMatrix);
EigenvalueDecomposition ed = new EigenvalueDecomposition(matrix);
eval1 = ed.getRealEigenvalues();// get charge weighted BCUT
LonePairElectronChecker lpcheck = new LonePairElectronChecker();
GasteigerPEPEPartialCharges pepe;
GasteigerMarsiliPartialCharges peoe;
lpcheck.saturate(mol);
double[] charges = new double[mol.getAtomCount()];
peoe = new GasteigerMarsiliPartialCharges();
peoe.assignGasteigerMarsiliSigmaPartialCharges(mol, true);
for (int i = 0; i < mol.getAtomCount(); i++) {
charges[i] += mol.getAtom(i).getCharge();
}
for (int i = 0; i < mol.getAtomCount(); i++) {
mol.getAtom(i).setCharge(charges[i]);
}counter = 0;
for (int i = 0; i < mol.getAtomCount(); i++) {
if (mol.getAtom(i).getSymbol().equals("H")) continue;
// diagvalue[counter] = 1.0; mol.getAtom(i).getCharge();
diagvalue[counter] = mol.getAtom(i).getCharge();
counter++;
}burdenMatrix = BurdenMatrix.evalMatrix(mol, diagvalue);
matrix = new Matrix(burdenMatrix);
ed = new EigenvalueDecomposition(matrix);
eval2 = ed.getRealEigenvalues();// get polarizability weighted BCUT
int[][] topoDistance = PathTools.computeFloydAPSP(
AdjacencyMatrix.getMatrix(mol));
Polarizability pol = new Polarizability();
counter = 0;
for (int i = 0; i < mol.getAtomCount(); i++) {
if (mol.getAtom(i).getSymbol().equals("H")) continue;
diagvalue[counter] = pol.calculateGHEffectiveAtomPolarizability(
mol, mol.getAtom(i), false, topoDistance);
counter++;
}
burdenMatrix = BurdenMatrix.evalMatrix(mol, diagvalue);
matrix = new Matrix(burdenMatrix);
ed = new EigenvalueDecomposition(matrix);
eval3 = ed.getRealEigenvalues();} catch (Exception e1) {
e1.printStackTrace();
}
System.out.printf("%s\t%.3f\t%.3f\t%.3f\t%.3f\t%.3f\t%.3f\n",
id,
eval1[0], eval1[eval1.length - 1],
eval2[0], eval2[eval2.length - 1],
eval3[0], eval3[eval3.length - 1] );
}
}static private class BurdenMatrix {
static double[][] evalMatrix(IAtomContainer ac, double[] vsd) {
IAtomContainer local = AtomContainerManipulator.removeHydrogens(ac);
int natom = local.getAtomCount();
double[][] matrix = new double[natom][natom];
for (int i = 0; i < natom; i++) {
for (int j = 0; j < natom; j++) {
matrix[i][j] = 0.0;
}
}/* set the off diagonal entries */
for (int i = 0; i < natom - 1; i++) {
for (int j = i + 1; j < natom; j++) {
for (int k = 0; k < local.getBondCount(); k++) {
IBond bond = local.getBond(k);
if (bond.contains(local.getAtom(i) ) &&
bond.contains(local.getAtom(j) ) ) {
if (bond.getFlag(CDKConstants.ISAROMATIC)){
matrix[i][j] = 0.15;
} else if (bond.getOrder() == CDKConstants.BONDORDER_SINGLE){
matrix[i][j] = 0.1;
} else if (bond.getOrder() == CDKConstants.BONDORDER_DOUBLE){
matrix[i][j] = 0.2;
} else if (bond.getOrder() == CDKConstants.BONDORDER_TRIPLE){
matrix[i][j] = 0.3;
}
if (local.getConnectedBondsCount(i) == 1 ||
local.getConnectedBondsCount(j) == 1) {
matrix[i][j] += 0.01;
}
matrix[j][i] = matrix[i][j];
// } else {
} else if( matrix[i][j] == 0.0 ){
matrix[i][j] = 0.001;
matrix[j][i] = 0.001;
}
}
}
}/* set the diagonal entries */
for (int i = 0; i < natom; i++) {
if (vsd != null) matrix[i][i] = vsd[i];
else matrix[i][i] = 0.0;
}
return (matrix);
}
}
}
固有値計算には、JAMAパッケージを用ゐていますね。
ちなみに、バグだと思われる場所を2ヶ所修正しましたので、
BCUTDescriptorクラスとは違う値が出力されると思います。
*1: Burden. Molecular Identification Number for Substructure Searches. J. Chem. Comput. Sci. (1989) 29, 225-227